近日,中医药创新研究院陈伟教授团队及合作者在生物信息学领域知名期刊《Bioinformatics》上发表了题为《DNA4mC-LIP: a linear integration method to identify N4-methylcytosine site in multiple species》的研究论文,提出了一种识别DNA胞嘧啶甲基化(N4-methylcytosine, 4mC)的新方法。
4mC作为重要的表观遗传修饰之一,在DNA修复、表达和复制等诸多生物过程中起着至关重要的作用。4mC位点的准确识别将有助于深入研究其生物学功能和机制。由于现有实验方法成本高、操作时间长等原因,亟需开发识别4mC的新方法。
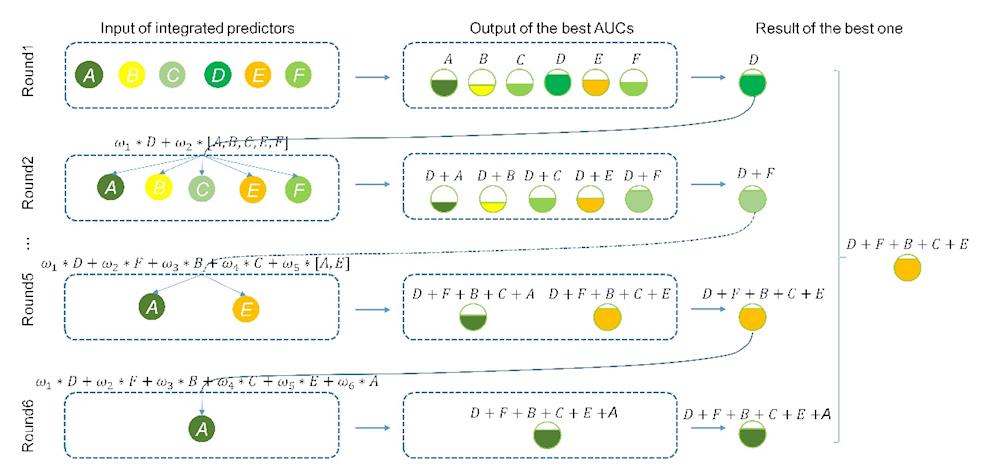
针对上述问题,本研究首先基于独立数据集对已有的4mC检测方法进行了系统性评价。然后使用集成学习方法,对当时已经报道的六种4mC检测方法进行加和集成,开发了一个准确率更高,性能更优越的新工具DNA4mC-LIP。为了便于应用,项目组还提供了该工具的在线服务http://i.uestc.edu.cn/DNA4mC-LIP/。DNA4mC-LIP的提出不仅弥补了实验检测4mC位点耗时费力的缺陷,也解决了现有理论方法准确性不高的不足。
创新研究院陈伟教授团队的唐强与电子科技大学康娟娟博士为共同第一作者,陈伟教授与电子科技大学生命科学与技术学院的黄健教授、林昊教授为本文的共同通讯作者。该研究得到了国家自然科学基金、威尼斯98488杏林学者青年教师创新基金等项目的支持。
自2019年以来,陈伟教授团队围绕核酸修饰在Briefings in Bioinformatics, Molecular Therapy-Nucleic Acids,Bioinformatics, Frontiers in Bioengineering and Biotechnology等期刊发表了相关学术论文10余篇。
文章链接:https://doi.org/10.1093/bioinformatics/btaa143。
(图、文/陈伟 编辑/科技处 张慧敏)


